6.4 差异性分析 EMP_diff_analysis
模块EMP_diff_analysis支持实验数据在不同分组条件下的差异性分析,涵盖多种统计检验方法,包括T检验、Wilcoxon检验、单因素方差分析(one-way.test)、Kruskal-Wallis检验、DESeq2、edgeR和limma等。该模块不仅支持多核并行运算以提升效率,还具备缓存快照功能(即:在重复使用相同参数进行计算时,系统将自动提取已缓存的结果,无需重新计算)。
6.4.1 传统统计学方法
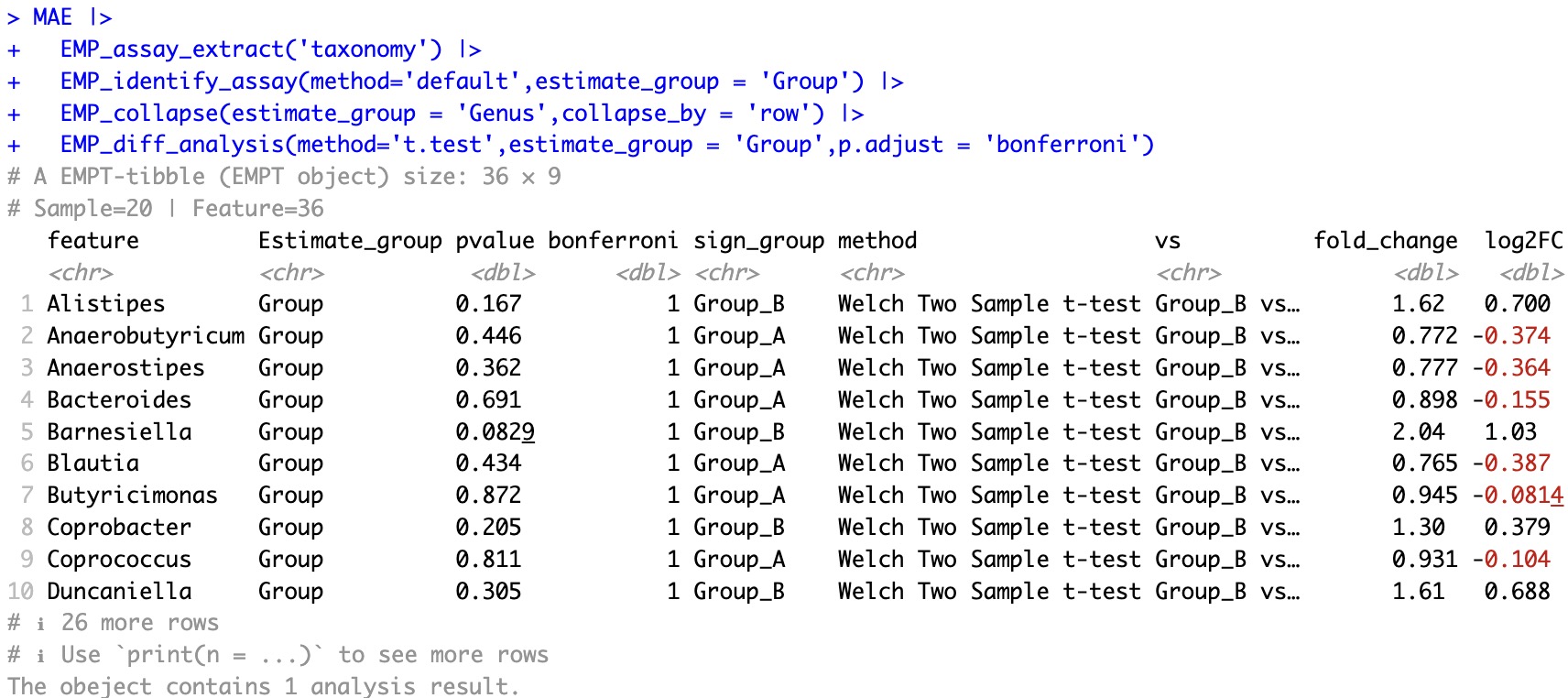
🏷️示例1:使用T检验计对属级别的核心微生物数据进行差异性分析,并对p值进行Bonferroni多重检验校正。
注意:
①在分组为两组的情况下,
②在分组超过两组的情况下,
③在计算两组间均值比值时,如果其中一个特征的值为0,则默认采用另一组对应特征的千分之一赋值,则这种情况下比值为1000或者0.001。
④如需要配对检验,请查看章节10.3 配对检验和干预队列的处理方案。
①在分组为两组的情况下,
sign_group显示的组别仅代表该特征在所述组别中的均值高于另一组别,但并不涉及统计检验。在本示例中,针对特征列的Alistipes,sign_Group为Group_B表示特征Alistipes在Group_B中的均值高于Group_A;结合pvalue列,T检验的p值为0.167,经Bonferroni校正后的p值为1,表明该特征在组间的差异不具有统计学意义。②在分组超过两组的情况下,
sign_group显示的组别仅代表该特征在所涉及组别中均值最高的分组,但并不涉及统计检验。pvalue列的p值小于0.05代表所涉及组别的均值不全相同,如果想要明确具体哪些分组的均值具有统计学差异,需要进行两两比较。③在计算两组间均值比值时,如果其中一个特征的值为0,则默认采用另一组对应特征的千分之一赋值,则这种情况下比值为1000或者0.001。
④如需要配对检验,请查看章节10.3 配对检验和干预队列的处理方案。
MAE |>
EMP_assay_extract('taxonomy') |>
EMP_identify_assay(method='default',estimate_group = 'Group') |>
EMP_collapse(estimate_group = 'Genus',collapse_by = 'row') |>
EMP_diff_analysis(method='t.test',estimate_group = 'Group',p.adjust = 'bonferroni')
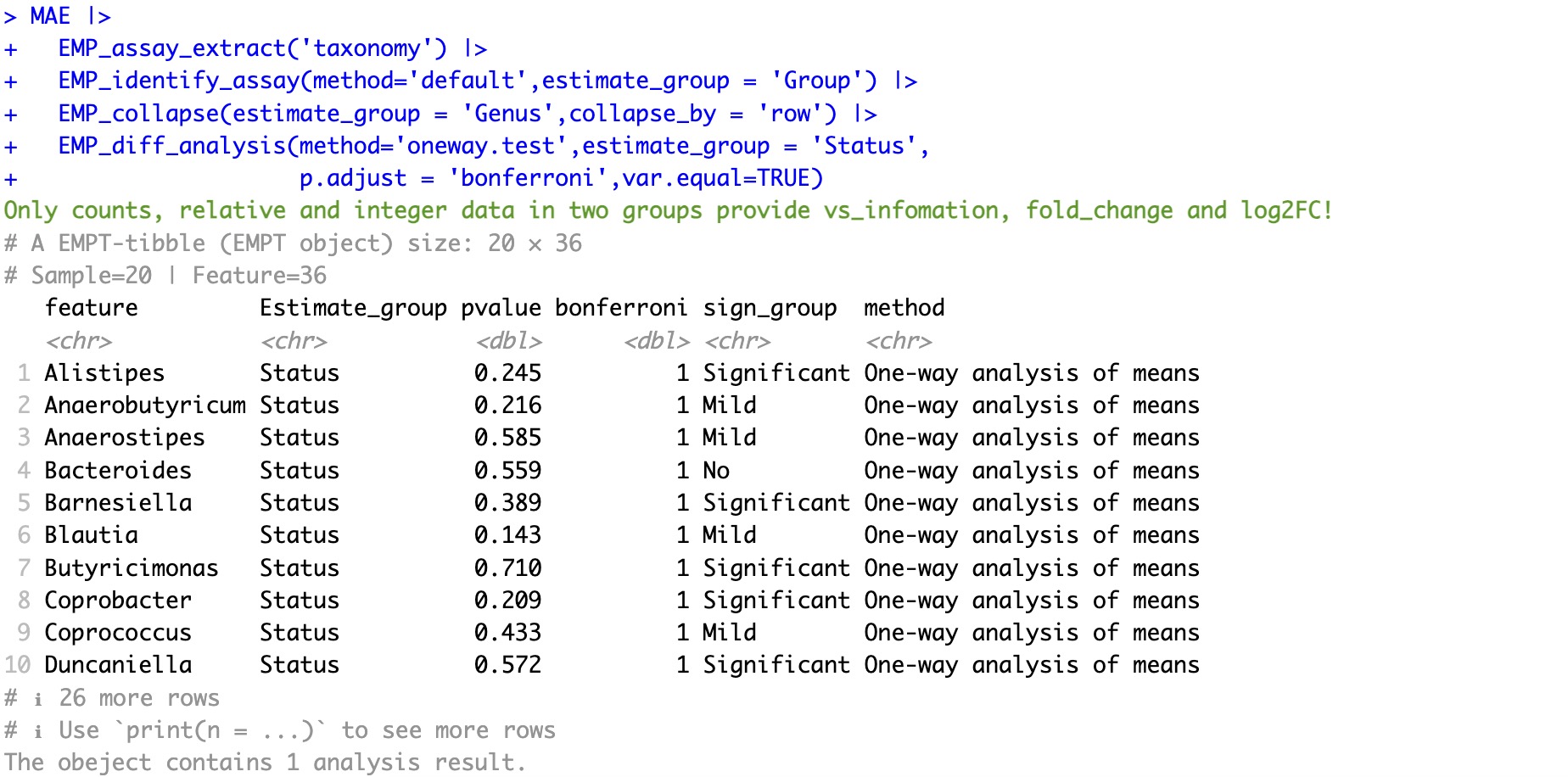
🏷️示例2:在分组超过两组的情况下,使用 oneway.test或者kruskal.test进行统计检验。
注意:
①在分组超过两组或者输入数据不是
②
①在分组超过两组或者输入数据不是
couts、relative、integer时,将无法提供fold_change、VSlog2FC等信息。②
EMP_diff_analysis继承了R内置stats包的方法,可以在模块内增加其参数进行调整。
MAE |>
EMP_assay_extract('taxonomy') |>
EMP_identify_assay(method='default',estimate_group = 'Group') |>
EMP_collapse(estimate_group = 'Genus',collapse_by = 'row') |>
EMP_diff_analysis(method='oneway.test',estimate_group = 'Status',
p.adjust = 'bonferroni',var.equal=TRUE)
6.4.2 DESeq2、edgeR和limma等方法
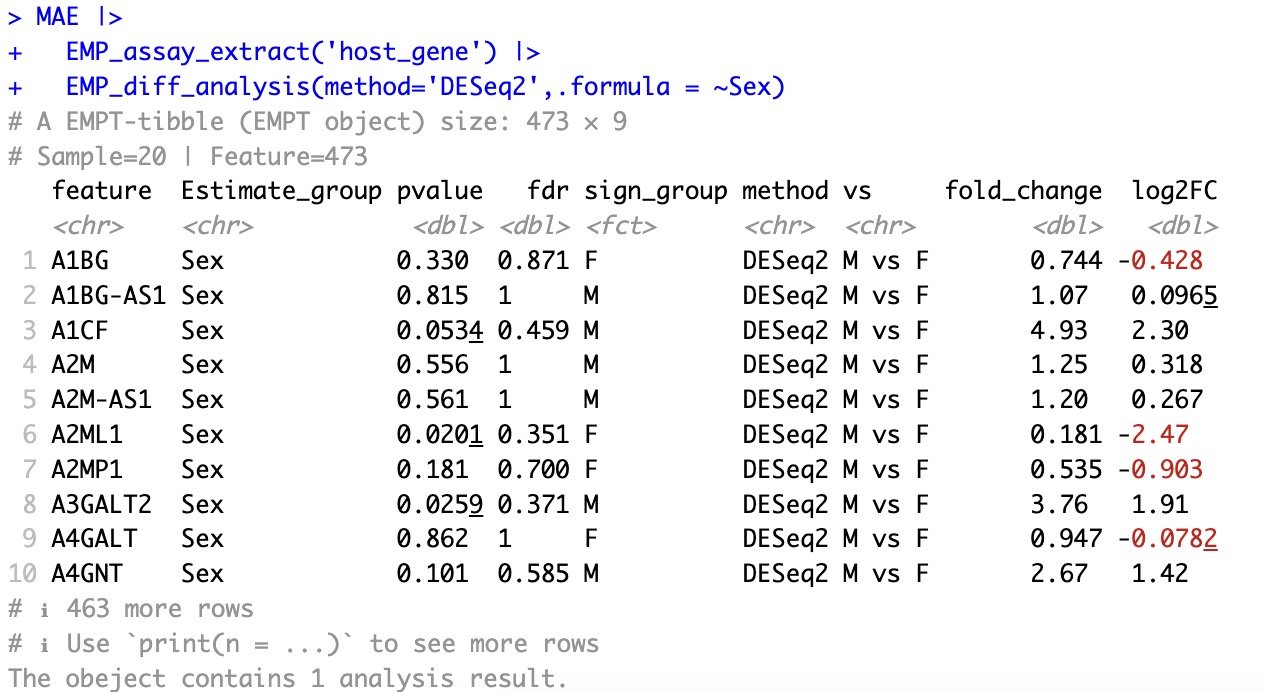
🏷️示例1:使用DESeq2对宿主基因数据集按照性别进行差异性分析。
注意:
在使用这些方法时,不再使用
在使用这些方法时,不再使用
estimate_group参数来指定分组信息,而是遵循所继承包的习惯语法.formula。
MAE |>
EMP_assay_extract('host_gene') |>
EMP_diff_analysis(method='DESeq2',.formula = ~Sex)
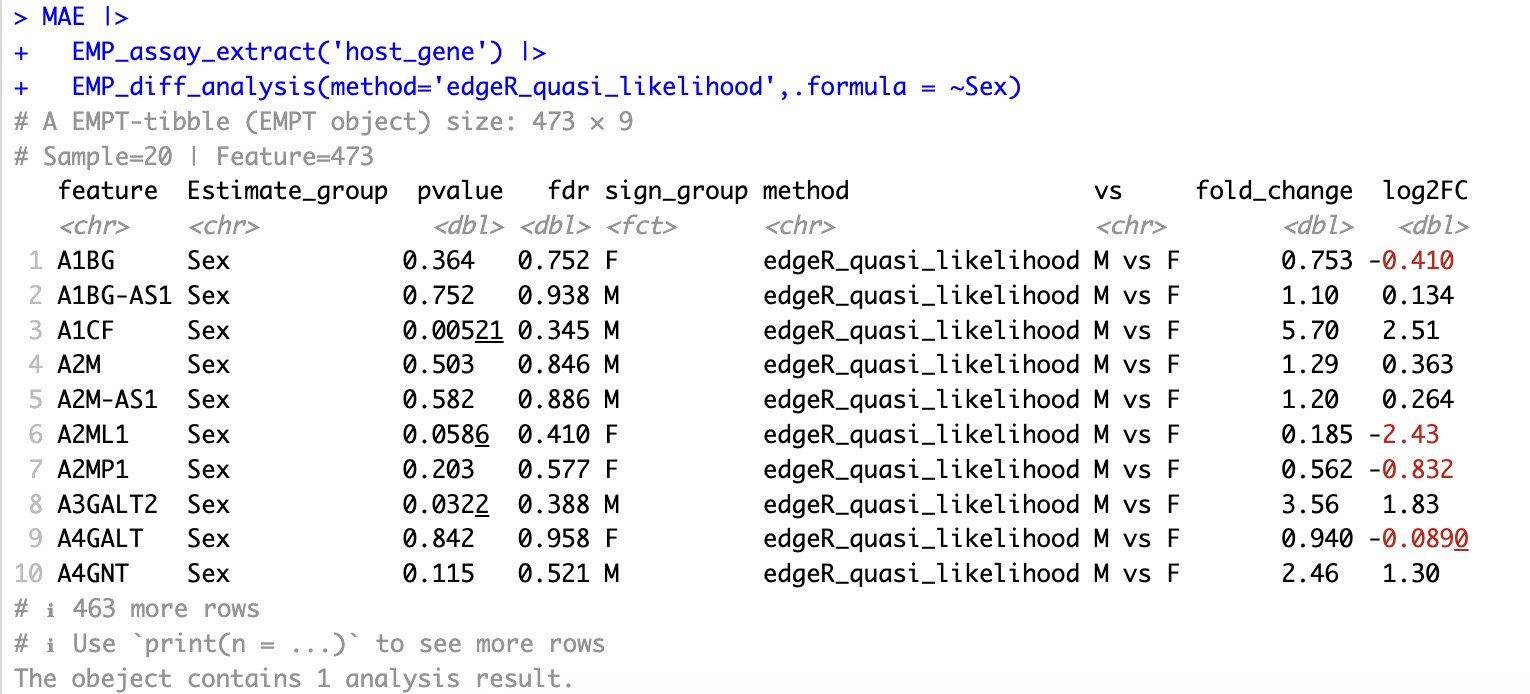
🏷️示例2:使用edgeR的几种模式方法。
MAE |>
EMP_assay_extract('host_gene') |>
EMP_diff_analysis(method='edgeR_quasi_likelihood',.formula = ~Sex)
MAE |>
EMP_assay_extract('host_gene') |>
EMP_diff_analysis(method='edgeR_likelihood_ratio',.formula = ~Sex)
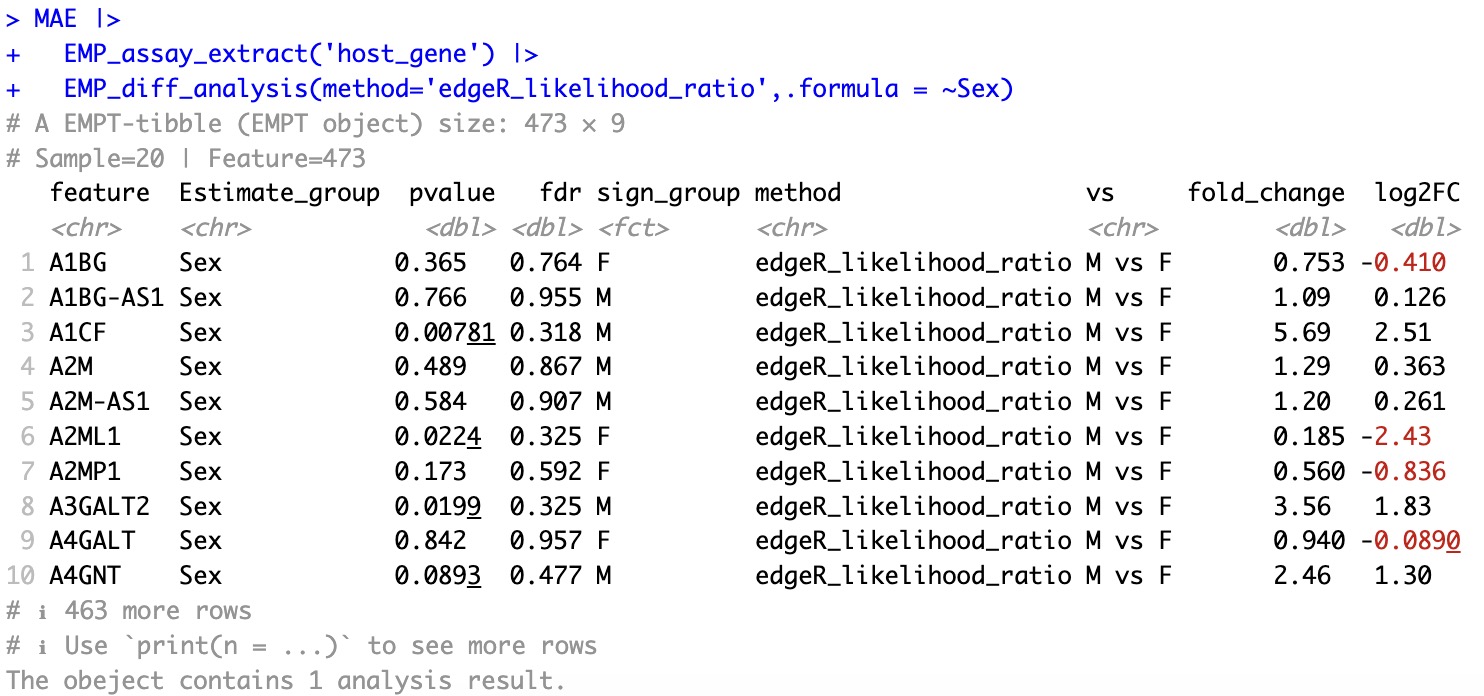
🏷️示例3:更多方法可以尝试。
MAE |>
EMP_assay_extract('host_gene') |>
EMP_diff_analysis(method='edger_robust_likelihood_ratio',.formula = ~Sex)
MAE |>
EMP_assay_extract('host_gene') |>
EMP_diff_analysis(method='limma_voom',.formula = ~Sex)
MAE |>
EMP_assay_extract('host_gene') |>
EMP_diff_analysis(method='limma_voom_sample_weights',.formula = ~Sex)
6.4.3 差异性分析结果过滤及可视化
模块EMP_filter可以帮助快速筛选分析结果,详细用法参见 模块EMP_filter帮助文档。
注意:
这里的参数
这里的参数
keep_result,详情请参看章节7.1.3
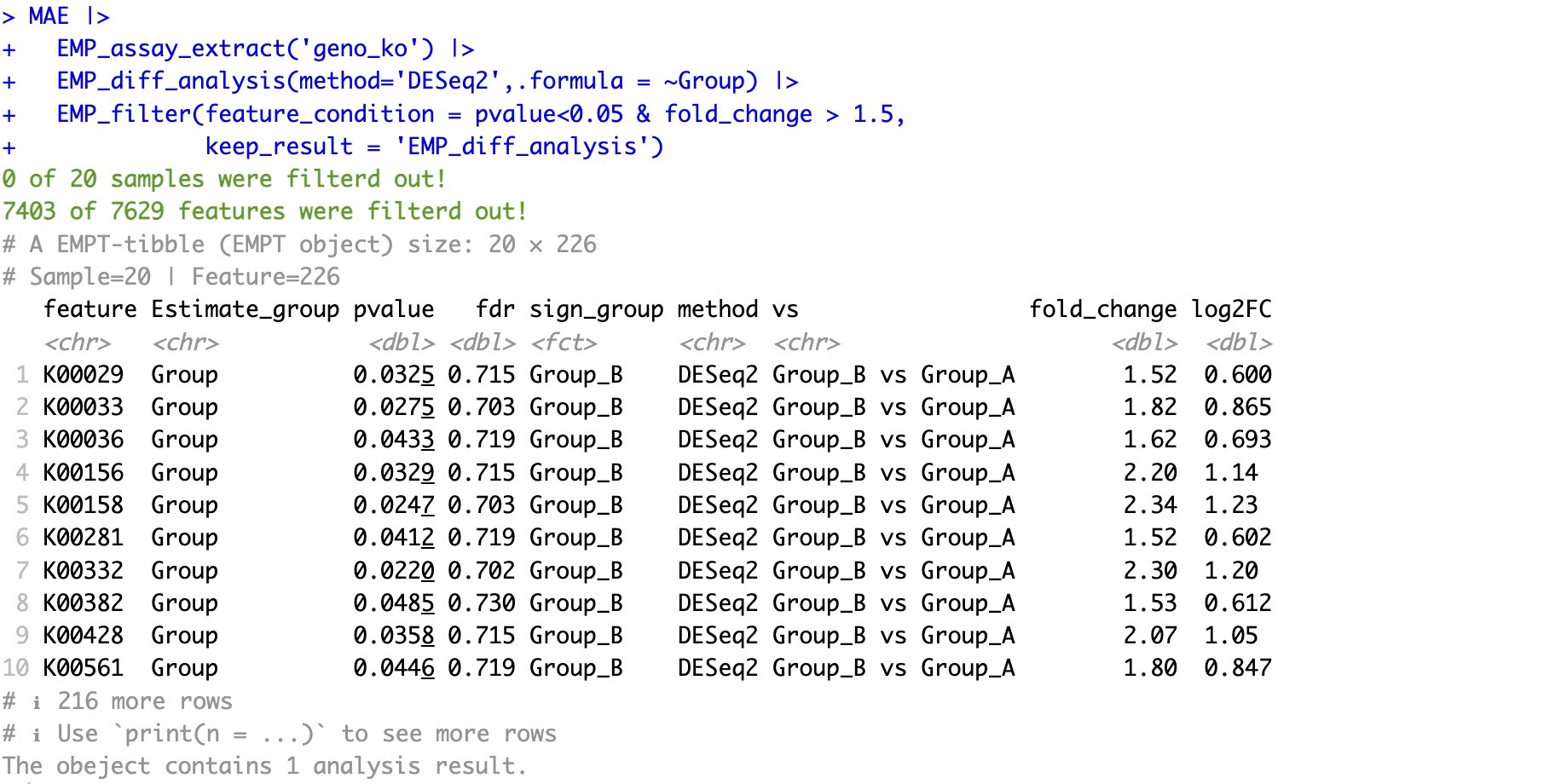
MAE |>
EMP_assay_extract('geno_ko') |>
EMP_diff_analysis(method='DESeq2',.formula = ~Group) |>
EMP_filter(feature_condition = pvalue<0.05 & fold_change > 1.5,
keep_result = 'EMP_diff_analysis')
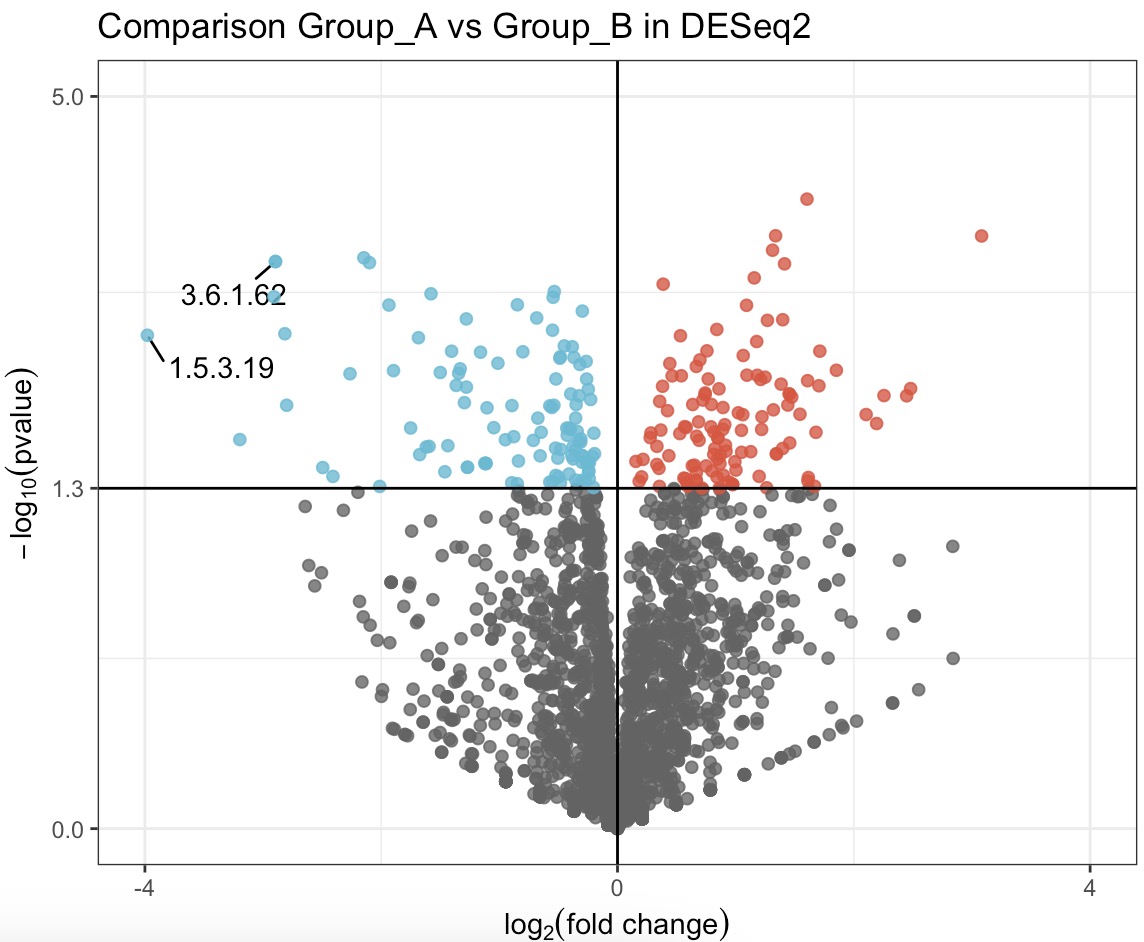
将差异性分析结果绘制成火山图。
注意:
在
在
EMP_volcanol_plot中增加show='html'可以提供可交互式图形,便于快速确定差异特征的具体信息。详见8.10 EMP_vocanol_plot。
MAE |>
EMP_decostand(experiment = 'geno_ec',method = 'integer') |>
EMP_diff_analysis(method='DESeq2',.formula = ~Group) |>
EMP_volcanol_plot(key_feature = c('3.6.1.62','1.5.3.19'),
min.segment.length = 0, seed = 42, box.padding = 0.5) ## Add arrow